せっかくだから Makie.jl で Flowcytometry の疑似データを3次元プロットしてみよう。
Makie.jl が、なかなか楽しいのです。「julia で、flowcytometry のデータを plot する」のデータを3次元に拡張して plot してみましょう。
インストールについては、@cometscome_phys さんの Juliaで綺麗なプロットを作る:Makie.jlのインストールと使い方 を見て下さい。
ぐりぐり回して、観察できるので楽しいです。
scatter(data, color=A_col) のところは、初めは自分の環境では、30秒以上かかります。2回目以降は、1秒かからず描画します。10^5 個のデータがありますが、かなり軽快に動きます。
using Makie, StatsBase, Distributions, ColorTypes
selection = rand(1:31000,100000);
data = vcat(
rand(Normal(),10000,2),
rand(Normal(3,0.8),10000,2),
rand(Normal(-2,2),10000,2).+[0 2],
rand(TDist(10),1000,2)
)[selection,:];
z_data = vcat(
rand(Normal(),10000),
rand(Normal(1,1),10000),
rand(Normal(1,1),3000).+4,
rand(Normal(1,1),7000),
rand(TDist(10),1000)
)[selection];
data=hcat(data, z_data);
# 色の指定をする関数です。
function Array_col(data::Array, col_map_name=:viridis::Symbol)
col_map = to_colormap(:viridis, 64);
A_col = [col_map[64] for i in 1:size(data,1)]
z = round.(data[:,3].*10) .- minimum(round.(data[:,3].*10))
step = quantile(z, 0.95)/64
# 条件に合う色を col_map[i] に書き変えていきます。
for i in 1:64
A_col[step.*(i-1).<=z.< step*i] .= col_map[i]
end
return A_col
end
A_col = Array_col(data);
scatter(data, color=A_col)
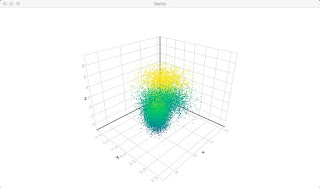

viridis の配色で、z軸の値で色を付けています。
おしまい。

